2-9序列比对和数据库搜索
基因组学
CNGB 中国国家基因库生命大数据平台
NCBI
NCBI开发有Genbank(基因序列)等公共数据库,提供Pubmed(生物医学文献检索)、BLAST(序列对比分析工具)、Entrez、OMIM、Taxonomy、Structure等工具,。
GENE检索
序列比对BLAST
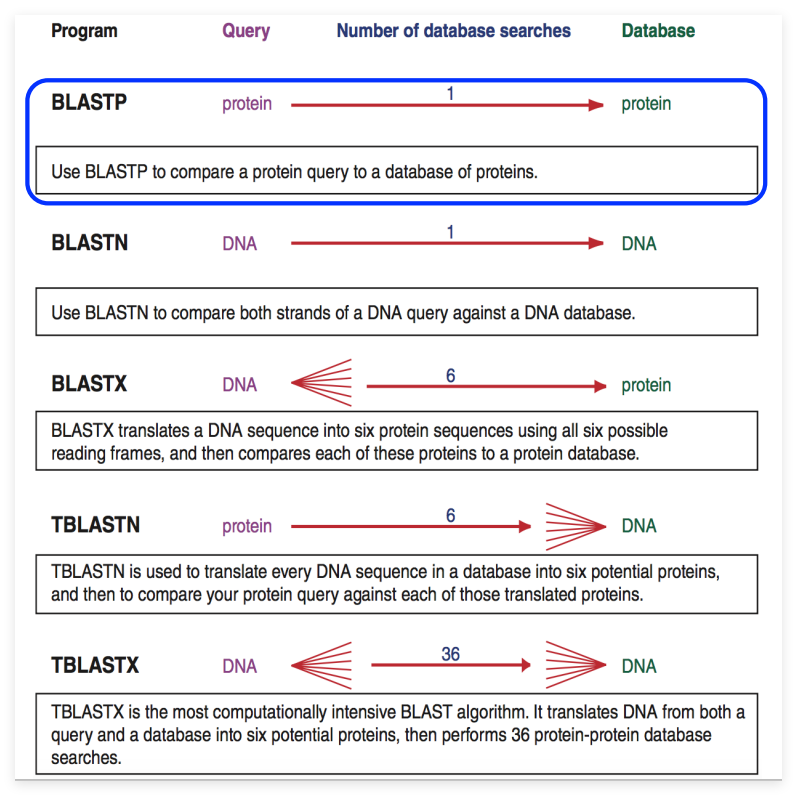
BLAST子程序说明(类型)
| 程序名 | 查询序列 | 数据库类型 | 方法或用途 |
|---|---|---|---|
| Blastp | 蛋白质 | 蛋白质 | 以蛋白质序列搜索蛋白质序列数据库 |
| Blastn | 核酸 | 核酸 | 以核酸序列搜索核酸序列数据库 |
| Blastx | 核酸 | 蛋白质 | 将核酸序列按6条链翻译成蛋白质序列后搜索蛋白质序列数据库 |
| tblastn | 蛋白质 | 核酸 | 以蛋白质序列搜索由核酸序列数据库按6条链翻译成的蛋白质序列数据库 |
| tblastx | 核酸 | 核酸 | 将核酸序列按6条链翻译成蛋白质序列后搜索由核酸序列数据库按6条链翻译成的蛋白质序列数据库 |
 |
|||
| blastn结果分析:Expect(E值)、Identities(一致性)、Gaps(缺失或插入)三项是评价blast结果的标准。E值接近零或者为零时,具体上就是完全匹配了;一致性:匹配上的碱基数占总序列长的百分数。 | |||
| 全局比对适用于功能相似序列(例如同源序列比对)的比对。 | |||
| 局部比对适用于查找子序列, 就是局部相似性很高的序列 |
多序列比对(clustalX)
蛋白质组学资源及数据库
ExPASy
ExPASy(Expert Protein Analysis System)是一个专门用于蛋白质和蛋白组学分析的生物信息学资源库。瑞士生物信息学研究所(Swiss Institute of ioinformatics, SIB)开发和维护。1993年上线。
String数据库
String是搜寻蛋白质之间相互作用的数据库。该数据库可应用于2031个物种,包含1380万种蛋白质之间的相互作用。
数据来源:实验数据、从PubMed文献中挖掘的结果和综合其它数据库数据、生物信息学的方法预测的结果。
代谢物通路分析
KEGG
代谢物注释和通路查找
HMDB(The Human Metabolome Database)
All articles in this blog are licensed under CC BY-NC-SA 4.0 unless stating additionally.
Comment



